文字列分解
遺伝子のヌクレオチドのリスト中のコドン(連続する3つのヌクレオチドのグループ)の相対頻度を調べる.
ヒト遺伝子"SCNN1A"のDNA配列を取得する.
In[1]:=
dnasequence = GenomeData["SCNN1A", "FullSequence"];StringPartitionを使って対応するコドンのリストを構築する.
In[2]:=
codons = StringPartition[dnasequence, 3];In[3]:=
Take[codons, 10]Out[3]=
この遺伝子中の各コドンの相対頻度を計算する.
In[4]:=
frequencies = N[Counts[codons]/Length[codons]];A,C,G,Tのヌクレオチドから形成可能なコドンは64ある.選択した遺伝子にこれらすべてが含まれている.
In[5]:=
frequencies // LengthOut[5]=
頻度が最も高い3つのコドンを求める.
In[6]:=
TakeLargest[frequencies, 3]Out[6]=
頻度が最も低い3つのコドンを求める.
In[7]:=
TakeSmallest[frequencies, 3]Out[7]=
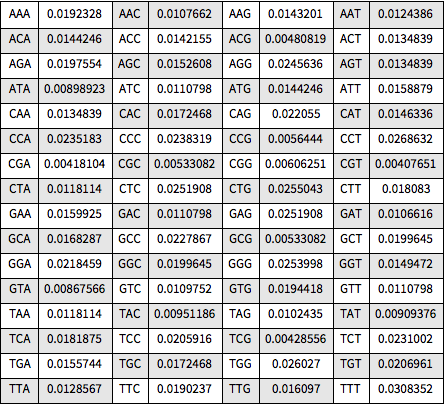
全相対頻度をGridを使って可視化する.
完全なWolfram言語入力を表示する
Out[8]=