Sequenzalignment und Clusteranalyse
Dieses Beispiel vergleicht evolutionär erhaltene (homologe) Gensequenzen zwischen verschiedenen Organismen unter Verwendung der Methoden Sequenzalignment und Clustering.
Das EME1-Gen weist Sequenzhomologien mit Vertretern unterschiedlichster Arten auf. Die Wolfram Knowledgebase liefert EME1-Sequenzdaten für Menschen, Schimpansen, Mäuse und Ratten, die sowohl von Wolfram|Alpha als auch von der Wolfram Language genutzt werden.
Hier ist zum Beispiel die EME1-Gensequenz für Mäuse.
Interpretieren Sie nun die EME1-Genentitäten für jeden der ausgewählten Organismen.
Von hier aus können Sie die Gensequenzen für jedes der ausgewählten Gene abrufen.
Betrachtet man die Anfänge der Sequenzanpassungen, so scheint es, dass die EME1-Gensequenzen für Mensch und Schimpanse viel ähnlicher sind als die für Ratten und Mäuse. Mensch und Maus EME1-Sequenzen erscheinen noch weniger ähnlich.
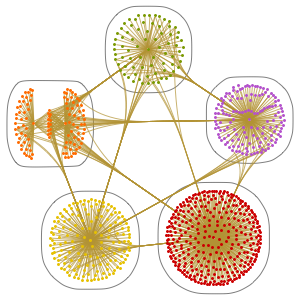
Mit dem Sequenz-Clustering können Sie sehen, wie ähnlich die gesamten Sequenzen tatsächlich sind. Berechnen Sie zunächst alle Ähnlichkeitsverhältnisse zwischen allen Sequenzen.
Man sieht starke Ähnlichkeiten zwischen den Chromosomen von Mensch und Schimpanse und mittlere Ähnlichkeiten zwischen Ratte und Maus.
Wenn Sie das Dendrogramm nehmen, können Sie die relative Dichtheit der Cluster sehen, die aus dieser Ähnlichkeit hervorgehen.
Betrachtet man die gesamte Ausrichtung, so erkennt man, dass menschliche EME1-Sequenzen nahezu perfekt mit Schimpansen-EME1-Sequenzen aligniert sind, während das EME1-Alignment zwischen Mäusen und Menschen eher willkürlich wirkt.
In der Musik können wir den Unterschied zwischen stärkerem und schwächerem Alignment hören, d.h., wo die Sequenzen größere bzw. kleinere Ähnlichkeit miteinander haben.
Hören Sie selbst, wie die Schimpansen-/Menschen-Alignment für EME1 klingt.
Vergleichen Sie das mit dem Klang des EME1-Alignment von Ratten und Mäusen.