Spleißstellen in einer DNA-Sequenz finden
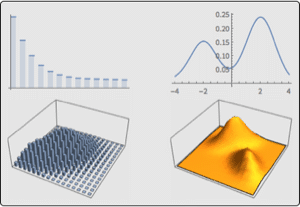
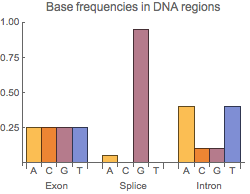
Eine DNA-Sequenz wird mit den Buchstaben A, C, G und T angegeben. Angenommen, eine Sequenz beginnt mit einem Exon, enthält eine Spleißstelle und endet mit einem Intron. Wenn die Exons eine einheitliche Basenzusammensetzung haben, die Introns weniger C und G aufweisen und das Spleißstellen-Konsensnukleotid mit der Wahrscheinlichkeit von 0,95 G ist, so sind die Häufigkeitsverteilungen wie folgt:
| In[1]:= |  X |
 |
| In[2]:= | X |
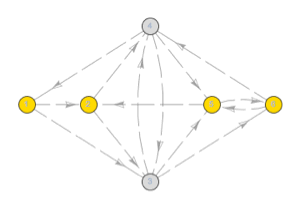
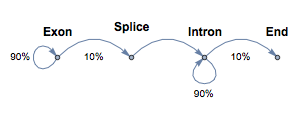
Der Automat nimmt Zustände für Exon (1), Spleißen (2), Intron (3) und Ende (4) an, mit den folgenden Übergangswahrscheinlichkeiten zwischen Zuständen:
 |
| In[3]:= | X |
Die Emissionen sind die Nukleotide A (1), C (2), G (3), T (4) oder Ende (5).
| In[4]:= | X |
| In[5]:= | X |
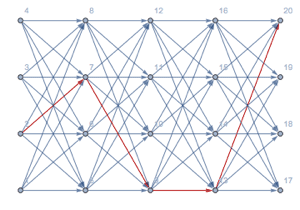
Finden Sie die wahrscheinlichste Nukleotidsubsequenz (Exon, Spleißen, Intron oder Ende).
| In[6]:= | X |
| Out[6]= |
Ermitteln Sie die Verbundwahrscheinlichkeit der Vorgänger-Nukleotidsequenz und der DNA-Sequenz.
| In[7]:= | X |
| Out[7]= |