Zerlegung von Strings
Untersuchen Sie die relative Häufigkeit von Codonen (eine Sequenz von drei Nukleobasen) in der Nucleotidenfolge eines Gens.
Ermitteln Sie die DNA-Sequenz des menschlichen Gens "SCNN1A".
In[1]:=
dnasequence = GenomeData["SCNN1A", "FullSequence"];Erstellen Sie mit StringPartition die entsprechende Liste der Codone.
In[2]:=
codons = StringPartition[dnasequence, 3];In[3]:=
Take[codons, 10]Out[3]=
Berechnen Sie die relative Häufigkeit jedes Codons in diesem Gen.
In[4]:=
frequencies = N[Counts[codons]/Length[codons]];64 mögliche Codone können aus den Nucleotiden A, C, G, T gebildet werden, und alle von ihnen kommen im ausgewählten Gen vor.
In[5]:=
frequencies // LengthOut[5]=
Finden Sie die drei häufigsten Codone.
In[6]:=
TakeLargest[frequencies, 3]Out[6]=
Finden Sie die drei am wenigsten häufigen Codone.
In[7]:=
TakeSmallest[frequencies, 3]Out[7]=
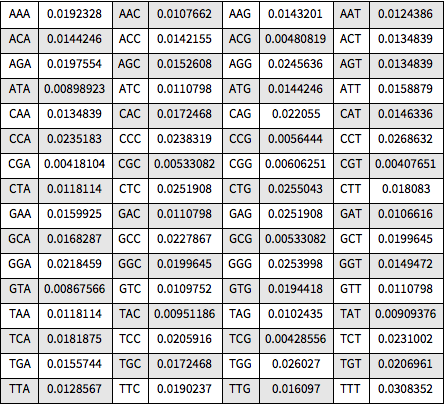
Visualisieren Sie alle relativen Häufigkeiten in einem Grid.
Den kompletten Wolfram Language-Input zeigen
Out[8]=